
Por Dra. Gabriela Olmedo (CINVESTAV, Irapuato)
Nota: estas notas sustituen la descripción de la visita que aparece en el volumen de las notas del Taller ("El Forense").
El experimento que vamos a realizar consiste en la deteccion de plantas transgenicas (plantas que fueron sumetidas a un tratamiento genetico).
Vamos a trabajar con muestras de ADN de plantas de maiz. Algunas de estas muestras han sido sometidas a un tratamiento genetico que le confiere tolerancia al aluminio. La contaminaci&oacvute;n por aluminio de ciertas plantas (como el maiz) es muy danina y causa fuertes pérdidas económicas. El tratamiento genetico que confiere a la planta del maiz la tolerancia al aluminio consiste en la introduccion de un gen (segmento de ADN) de una bacteria llamada citrato sintasa (CS).
El proposito de nuestro experimento es detectar las muestras de ADN de plantas de maiz que fueron sumetidos a este tratamiento genetico.
Para eso, vamos a utilizar una de las tecnicas fundamentales de la ingeneria genetica llamada PCR. Trabajarás con un poco de ADN de tejido de hoja de maiz y lo vas a someter a PCR utilizando oligos específicos para el gen CS y después analizarás tu resultado por electroforésis en gel de agarosa...
Está claro, ¿verdad?
Todos los seres vivos tienen ADN. El ADN es la molécula que tiene toda la información genética de cada individuo. Si eres hombre o mujer, güero(a) o moreno(a), alto(a) o bajo(a), se lo debes a tu ADN. Hoy en día entendemos bastante bien cómo se sintetiza el ADN y cómo parte de su información se utiliza para la síntesis de proteínas en cada célula. Entender todas estas cosas es el campo de acción de la llamada biología molecular. Una técnica grandiosa, fabulosa y utilísima en biología molecular es la "reacción en cadena de la polimerasa", mejor conocida como PCR (polymerase chain reaction). Pero antes de entrar en esto, déjame primero explicarte un poco qué es el ADN y qué es la "replicación".
La estructura de la doble cadena de ADN.
En los humanos y en muchos otros organismos superiores, la molécula de ADN consiste de dos cadenas enrolladas una sobre la otra. Cada cadena es un arreglo lineal de unidades repetidas llamadas nucleótidos, compuestos de un azúcar, fosfato y una base nitrogenada (Fig. 1). Hay 4 bases en el ADN: adenina (A), timina (T), citosina (C), y guanina (G). De aquí en adelante sólo hablaremos de A, G, C o T para referirnos a ellas. El orden de las bases es lo que conocemos como la secuencia del ADN y es la clave para almacenar la tremenda cantidad de información que necesita un organismo. Toda la información de ADN en una célula humana se guarda en 3,000,000,000 de pares de bases.
Con 27 letras del alfabeto se pueden hacer ¿Cuántas palabras? ¿Qué tan largas? (Parangaricutirimícuaro=22 letras). Si cuatro letras te parecen poco (A,G,C y T), prueba a arreglarlas en cualquier combinación y en cualquier tamaño, desde 70 ¡hasta 4000!
Reglas para formar la doble cadena de ADN
Para formar la doble cadena del ADN hay unas reglas estrictas:
1.Las dos cadenas que forman la doble cadena aparecen con una en un sentido y la otra va en el otro sentido. Las cadenas empiezan con un fosfato (P) y terminan con un hidroxilo (OH).
2.La A de una cadena sólo puede aparearse con la T de otra cadena, y lo mismo para la C, que sólo puede aparearse con la G.
Por ejemplo, segun estas reglas, una cadena
P-AAAACCCCTTTTGGGG-OH
sólo puede formar una cadena doble con una cadena
OH-TTTTGGGGAAAACCCC-P
(esta sería una cadena complementaria a la primera).
Ahora tú deduce la secuencia de la cadena complementaria del siguiente ADN:
P-TTAGCCCCCGGGTTAAA-OH
(El apareamiento de bases involucra la formación de puentes de hidrógeno, esto es, que la unión de pares de bases complementarios es energéticamente favorable).
Cada vez que una célula se divide, todo su ADN (su "genoma") se duplica. Durante la división la doble cadena se va separando y cada cadena dirige la síntesis de una cadena complementaria. Para esto usa nucleótidos A, G, C, y T que tiene a su disposición, que se "adhieren" siguiendo las reglas anteriores. Cada célula recibe entonces un ADN formado por la cadena viejita que sirvió como molde, unida a la nueva que se formó sobre ésta. De este modo se forman cadenas de ADN idénticas a la anterior y se conserva la información genética.
El proceso de replicación no es espontáneo. Requiere de múltiples enzimas. Podemos simplificarlo y hacerlo in vitro con tan sólo ADN polimerasa, primers y nucleótidos.
¿Cuáles primers, si ni se habían mencionado?
Ya, sé, espérense...
La ADN polimerasa es una enzima muy particular que requiere de un "primer". La ADN polimerasa tiene poca iniciativa: no inicia la síntesis de ADN si no tiene un pedacito ya comenzado (un adelanto). Este pedacito se llamará en adelante "primer" y marca el lugar desde el cual la ADN polimerasa simplemente seguirá, a toda velocidad y sin parar, añadiendo nucleótidos complementarios a la secuencia de la cadena molde. Sin discutir el porqué de esta actitud de la polimerasa, pasemos a lo del PCR y veremos las implicaciones que esto tiene para la técnica en cuestión.
Figura 1.

En la doble cadena las dos cadenas van en sentido opuesto y con cada base dispuesta en contraposición (formando puentes de hidrógeno) de exactamente su base complementaria: A - T y G - C. La polimerización de una nueva cadena complementaria se hace usando como molde cada una de las cadenas; éstas se abren y la ADN polimerasa las usa como molde para sintetizar la cadenas complementarias. Si la ADN polimerasa no recibe un "adelanto" (primer) no trabaja.
¿Qué es el PCR?
Mediante la técnica de PCR un segmento definido de ADN puede ser copiado repedidamente in vitro. Esto resulta en la amplificación de secuencias que originalmente existían en un muy reducido número.
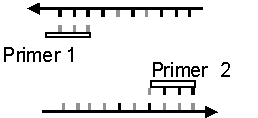
La clave de este método está en la enzima ADN polimerasa. Esta es una enzima que puede utilizar una cadena de ADN para polimerizar una cadena complementaria a ésta. Sin embargo, esta enzima no puede empezar de cero. Necesita un pequeño segmento de ADN que esté ya unido al ADN que va a copiar, esto es, necesita un primer. Bien, si tengo un fragmento de ADN de doble cadena y dos primers distintos y cada uno complementario a una de las cadenas del ADN molde, puedo hacer que cada uno de los primers sea el iniciador de la replicación de su cadena molde, como se muestra a continuación:
A. Abro la cadena doble del ADN y permito que cada cadena se acople a su primer complementario.
B. Añado polimerasa y oligonucleótidos y dejo se se copie in vitro cada cadena.
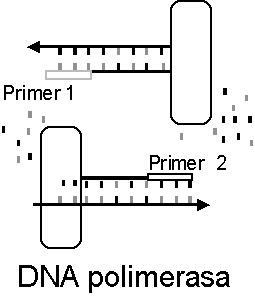
Y ahora viene la amplificación:
C. Cada cadena sintetizada servirá a su vez como molde para copiar una nueva cadena, que a su vez servirá para sintetizar otra cadena, que a su vez ...

De aquí el nombre de reacción en cadena.
A medida que procede la amplificación, la secuencia de ADN se duplica en cada ciclo. Después de 30 ciclos, la amplificación teórica lograda será de 1 000 000 000.
¿Cómo se lleva a cabo el PCR?
Lo primero es separar la doble cadena de ADN, y esto se logra rompiendo por calor los puentes de hidrogeno que las mantienen unidas. Las secuencias de 20 a 30 bases que sirven como primers se diseñan (20-30 nucleotidos) en base a la secuencia que ya conocemos en el ADN que deseamos amplificar y se sintetizan in vitro. Uno de los primers será complementario a una de las cadenas, y a una secuencia localizada al principio de lo que queremos amplificar, y el otro oligonucleótido será complementario a la cadena opuesta y estará al final de la región de interés.
Al utilizar calor para abrir la doble cadena de ADN en cada ciclo, es necesario emplear una ADN polimerasa que resista este tratamiento. Para esto se utiliza la enzima Taq polimerasa, aislada precisamente de una bacteria que habita en aguas térmicas, Thermus aquaticus. Así, a 90ºC se abre la cadena, a 50-60ºC se permite que se una el primer complementario, y a 72ºC se permite la polimerización. Además, todos los ciclos se realizan de manera automatica en máquinas llamadas termocicladores. .
En una reacción de PCR, una cantidad mínima de ADN se añade a un tubo pequeño que contiene una solución de sales con ADN polimerasa, los primers apropiados, los 4 oligonucleótidos y el cofactor. La mezcla se lleva a los ciclos de replicación mencionados. Una vez hecha la amplificación, ya sólo queda observar el ADN resultante por electroforesis. Aquellos que no pudieron ni pronunciar "electroforesis", pasen a esa sección.
¿Cómo saber si realmente funcionó la reacción en cadena de la polimerasa?
No podemos ver el ADN a simple vista, pero podemos ver y manipular MUCHAS moléculas de ADN que hemos sintetizado, si las ponemos en un gel y luego los teñimos con un colorante especial para ADN. Si bien la amplificación de ADN por PCR es impresionante, es importante recordar que la amplificación es selectiva; sólo el segmento de ADN flanqueado por los primers se amplificó exponencialmente. El resto de ADN no se amplifica y queda invisible en el gel.
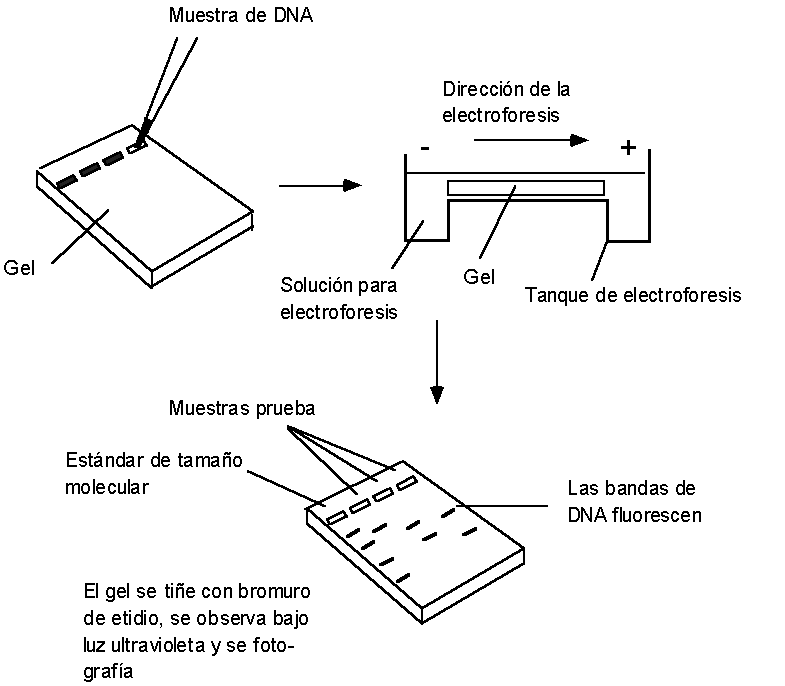
El gel se prepara dentro de un molde con agarosa y queda ni más ni menos que como una gelatina (ver la Fig. 2). Si dejamos algunos huecos en la parte superior del gel (pozos) los podemos llenar con el ADN que queremos observar. Aquí es donde hablamos de ELECTROFORESIS. El gel de agarosa está sumergido en una solución de sales (ésta permite conducir electricidad) dentro de una caja con un electrodo a cada extremo. Aplicamos un voltaje de manera tal que en el extremo del gel en donde está el ADN quede una carga negativa y al otro extremo quede positiva; el ADN, que tiene una carga negativa se moverá a través del gel hacia la carga positiva. Las moléculas más pequeñas podrán sortear más fácilmente los obstáculos del gel (imagínate una esponja) y avanzarán más rápidamente. El resultado final es que las moléculas de ADN se separan por tamaño (los de adelante (moléculas más pequeñas) corren mucho y los de atrás se quedarán (moléculas más grandes), trás, trás ). Este gel ahora se sumerge en una solución de un compuesto fluorescente llamado bromuro de etidio, el cual se intercala al ADN y nos permitirá verlo si utilizamos luz ultravioleta. Podemos ahora fotografiar este gel y utilizar la fotografía para analizar los resultados. Si usamos fragmentos de ADN de tamaño conocido como estándar, podremos estudiar y determinar el tamaño de las moléculas por comparación con el estándar.
Figura 2. Técnica de electroforésis
Uso de PCR para la detección de un gen "introducido"
Existen múltiples aplicaciones para el PCR, pero una de las más importantes, y de la que nos ocuparemos en nuestro experimento es la de detección. El gran poder de amplificación del PCR puede utilizarse para detectar la presencia de material genético que nos interese, ya sea para ver si está alterado, ver la presencia del material genético de un patógeno, como en el caso de las infecciones bacterianas o virales, o como en este caso, la presencia de un gen que normalmente no existe en el organismo, pero que se ha introducido por medio de técnicas de ingeniería genética. El PCR es flexible y fácil de realizar en muestras de distinata naturaleza y ha sido utilizada con gran éxito en la investigación y diagnóstico de infecciones o de enfermedades hereditarias.
Para que lo crean: La probabilidad de encontrar una secuencia de ADN definida de tan sólo cuatro bases es:
1/4 (primera posición) X 1/4 (segunda posición) X 1/4 (tercera posición) X 1/4 (cuarta posición) (1/4 viene de que son 4 posibles bases AGCT) = 1/256. Encontraremos esta secuencia tal vez cada 256 pares de bases.
Para 12 pares de bases: 1 vez cada 16,777,216 pares de bases
Para 20 pares de bases: 1 vez cada 90,949,000,000,000 pares de bases
En el experimento detectaremos el gen bacteriano de la citrato sintasa por la técnica de PCR. Se espera que este gen le de una gran ventaja al maiz. Como ya se mencionó, el PCR es una técnica poderosa que tiene gran importancia para la detección y caracterización de agentes infecciosos, incluidos los virus de plantas. En este experimento, utilizaremos un extracto crudo de ácidos nucleicos de hojas de maiz en un ensayo de PCR para detectar la presencia del gen CS.
Se utilizarán ADNs provenientes de hojas de plantas de maiz tranformadas y como control se usará ADN de hojas de maiz no transformado (C. negativo) y ADN de una planta que se sabe esta transformada (C. positivo). Tu tendrás que averiguar si las muestras que recibiste corresponden o no a maiz transgénico.
Material de laboratorio
Reactivos
La actividad:
1. El PCR se hará en un volúmen total de 50 µl de la siguiente manera: Tubo C+, ADN del control positivo Tubo C-, ADN del control negativo Tubo No. X, ADN de maiz a probar Tubo Y, ADN de maiz a probar Mezcla de PCR (NTPs, buffer y Taq pol, 13 µl primers) Extracto de hoja (ADN) 5 µl Agua 32 Total 50 µl
2. Una vez hechas las mezclas, se les pondrá dos gotas de aceite mineral para prevenir su evaporación y se mantendrán en hielo. A las 11:15 a más tardar los llevan al termociclador de Juan Pablo Martínez .
3. Los tubos serán incubados en un termociclador con el siguiente programa: un paso a 94 °C , 5 min seguido por 30 ciclos a 94°C 1 min, 62 oC 30 s, 72°C 30 s (un paso final de extension a 72 °C 7 min).
4. Una vez finalizado el ciclo de PCR, verificar la presencia del fragmento de amplificación esperado mediante análisis de electroforesis de 5-10 ml de la mezcla de PCR en un gel de agarosa al 1.6%. Carril 1: marcador de PM (1000 pb), carril 2, tubo C+, carril 3, tubo C-, carriles 4 y 5, muestras X y Y. Correr, teñir y fotografiar.
Resultados y Discusion
Examina la fotografía de tu gel. Comencemos por el marcador estándar. ¿Cuantas bandas ves? ¿Cuáles moléculas son mayores, las de "arriba" o las de "abajo"? .
¿Puedes explicar los resultados de los experimentos de PCR? Posibilidad 1. Se detecta una banda sólo en uno de los carriles. ¿Puedes saber si la banda que observas realmente corresponde a la amplificación por PCR ¿Tiene el tamaño esperado? ¿Que querría decir este resultado?
Posibilidad 2 (escribe tu resultado si es diferente al 1 y comenta al respecto)
10:00 Llegada al CINVESTAV. Charla introductoria por Aileen O´Connor.
10:30 Ubicación de grupos en los diferentes laboratorios. Comienza montaje de PCR según protocolo.
11:15 Comienza reacción de PCR en termociclador
11:45-Recorridos. Aileen O´Connor
1:30 COMIDA
2:30 Drs. Alba Jofre y Laura Silva: Plantas transgénicas y bioseguridad
3:30 Preparación de mini-gel de agarosa. Correr electroforesis con las muestras de la reacción de PCR Convivencia con integrantes del laboratorio asignado para conocer un poco del trabajo que allí realizan. Teñir gel y tomar foto del resultado.
5:00 Reunión para discutir resultados.
1. Andrés Cruz, Pedro García
2. Juan Pablo Martínez, Juan Carlos Ochoa
3. Alejandro Blanco
4. Víctor Olalde
5. Edmundo Lozoya, Saul Fraire
6. Plinio Guzmán, Jaime Bravo
7. Gabriela Olmedo, Jorge Ramírez
8. Bety Jiménez, Guillermo Corona
9. José Ruiz , Claudia León
10. Neftalí Ochoa, Héctor Górdon
ESCRITO POR: Gabriela Olmedo
DIRECCION TECNICA: Aileen O`Connor, Eva I. Martínez y Francisco Ayala
TECNICAS: PCR y electroforésis.
REPARTO: 10 laboratorios e igual número de estudiantes de posgrado y auxiliares de laboratorio.
RECORRIDOS: Aileen O´Connor